tar_make(script = "code/psp/_targets.r")Parcelles de Suvi Permanent
Permanent Sampling Plots
Reproductibilité
Pour reproduire ce document dynamique, incluant les analyses descriptives et graphiques :
- Cloner le dépot https://framagit.org/stafor/stafor.frama.io.
- Installer
quarto - Dans , installer et charger le package targets
- Lancer :
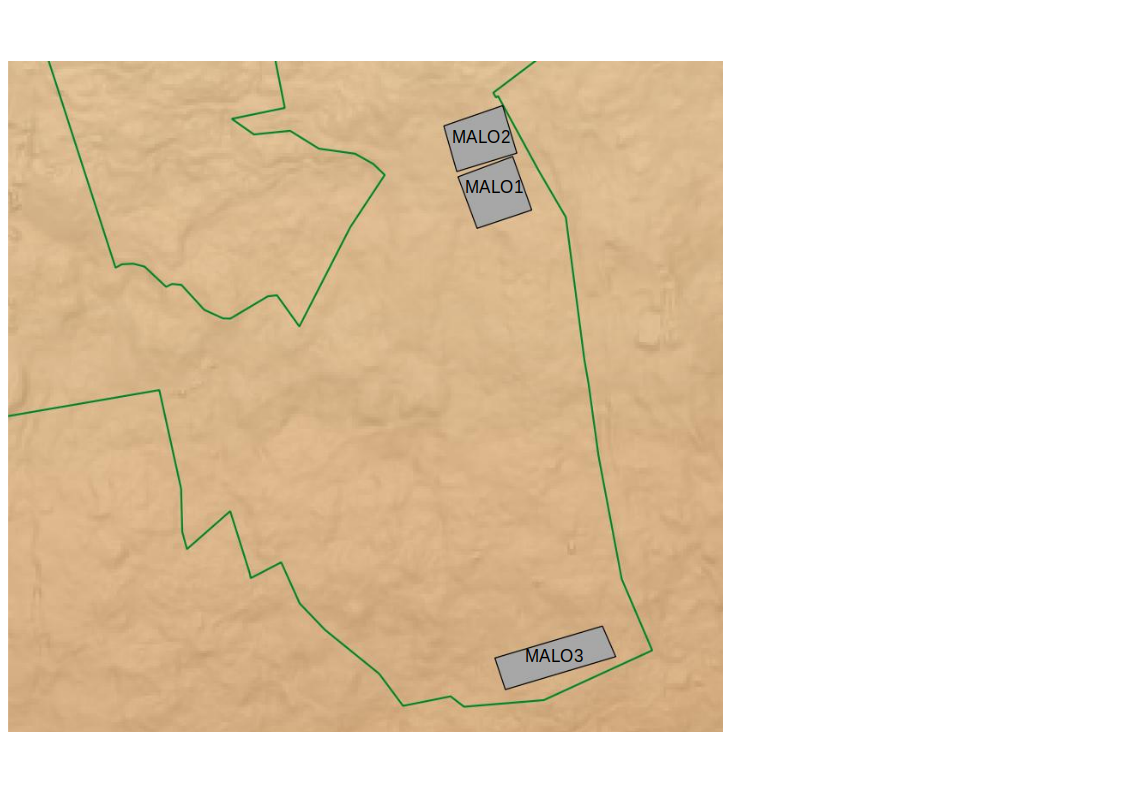
Exploration
Richesse spécifique
- Nombre de taxons total:
[1] 85[1] 85- Richesse spécifique par parcelle
| MALO1 | MALO2 | MALO3 |
|---|---|---|
| 44 | 67 | 56 |
Effectifs
Individus vivants par parcelle par année
Dénominations des années d’inventaire pour comparaison
| plot_id | T | annee |
|---|---|---|
| MALO1 | T0 | 2005 |
| MALO1 | T1 | 2012 |
| MALO1 | T2 | 2090 |
| MALO1 | T3 | 2099 |
| MALO2 | T0 | 2012 |
| MALO2 | T1 | 2017 |
| MALO2 | T2 | 2022 |
| MALO3 | T0 | 2003 |
| MALO3 | T1 | 2007 |
| MALO3 | T2 | 2014 |
| MALO3 | T3 | 2019 |
| MALO3 | T4 | 2024 |
Vivants par parcelle par année (0 : non concerné)
Effectifs par classe de DBH
| plot_id | T | annee | [0,1) | [1,4) | [4,8) | [8,1e+04] |
|---|---|---|---|---|---|---|
| MALO1 | T0 | 2005 | 0 | 3491 | 733 | 1106 |
| MALO1 | T1 | 2012 | 0 | 3053 | 637 | 1111 |
| MALO1 | T2 | 2090 | 0 | 0 | 0 | 685 |
| MALO1 | T3 | 2099 | 0 | 0 | 0 | 759 |
| MALO2 | T0 | 2012 | 43 | 3852 | 971 | 1244 |
| MALO2 | T1 | 2017 | 23 | 2763 | 285 | 502 |
| MALO2 | T2 | 2022 | 19 | 2103 | 643 | 1126 |
| MALO3 | T0 | 2003 | 0 | 0 | 0 | 898 |
| MALO3 | T1 | 2007 | 0 | 100 | 9 | 886 |
| MALO3 | T2 | 2014 | 0 | 2250 | 541 | 1145 |
| MALO3 | T3 | 2019 | 5 | 1919 | 523 | 1629 |
| MALO3 | T4 | 2024 | 3 | 1562 | 460 | 1558 |
- Hétérogénéité des inventaires …
- Pour le recrutement, analyse limitée aux individus > 4 en T3 (cf. MALO2 en 2016)
- Nombreux individus < 1 cm sur MALO3 … origine ??
Abondance des espèces
Toutes parcelles confondues en T3 :
- Grosses variations d’effectifs dues aux différences de limites d’inventaire
- 42 espèces à plus de 10 individus
- 5 espèces à 0 en T3
- Quelques corrections à apporter, cf PSYJAM ? (ah ces M2…)
| MALO1 | MALO2 | MALO3 | |
|---|---|---|---|
| T0 | 5330 | 6110 | 898 |
| T1 | 4801 | 3573 | 995 |
| T2 | 685 | 3891 | 3936 |
| T3 | 759 | 0 | 4076 |
| T4 | 0 | 0 | 3583 |
Figures
Warning in scale_x_log10(): log-10 transformation introduced infinite values.Warning: Removed 29810 rows containing non-finite outside the scale range
(`stat_bin()`).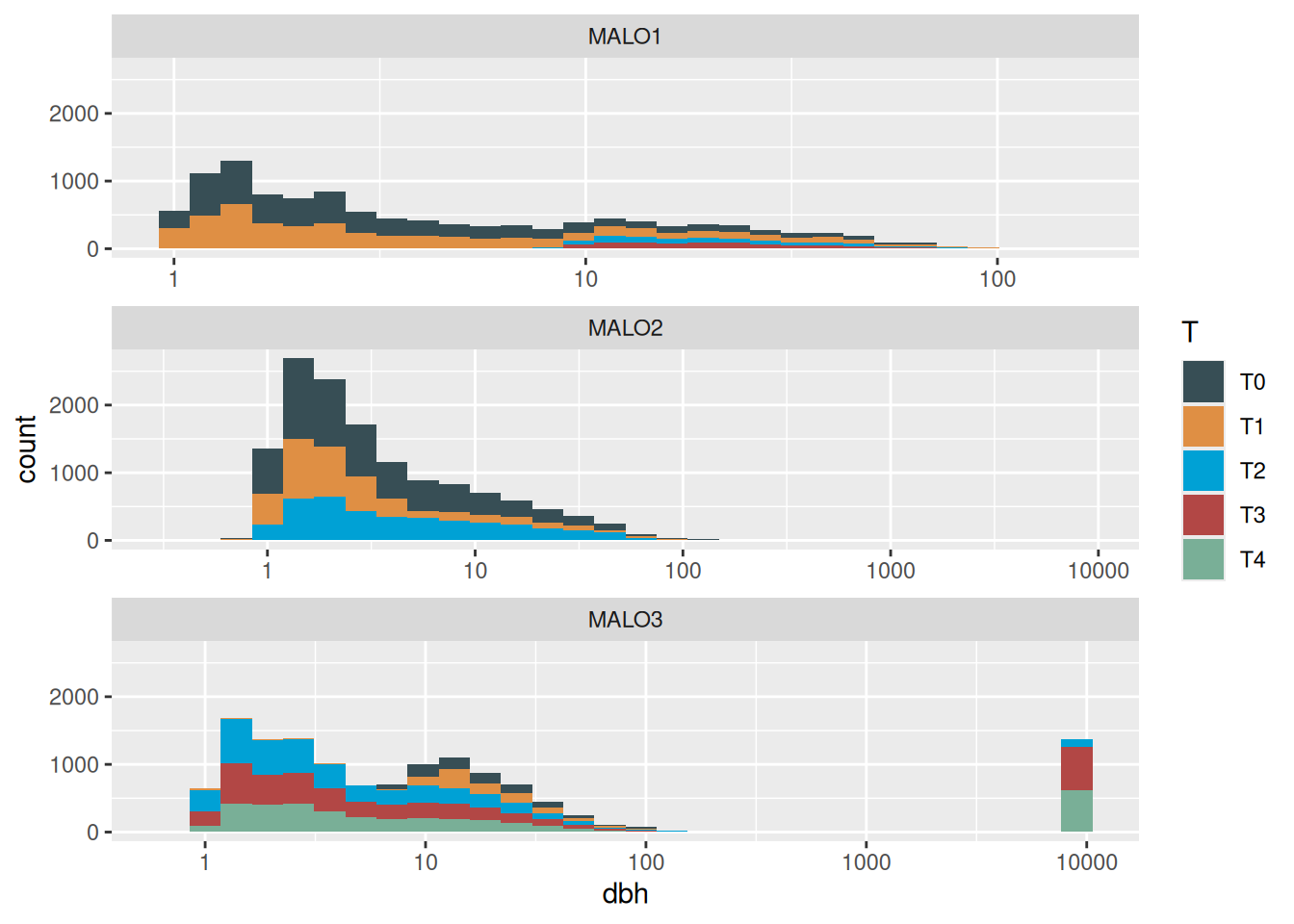
Warning in scale_x_log10(): log-10 transformation introduced infinite values.Warning: Removed 29810 rows containing non-finite outside the scale range
(`stat_density()`).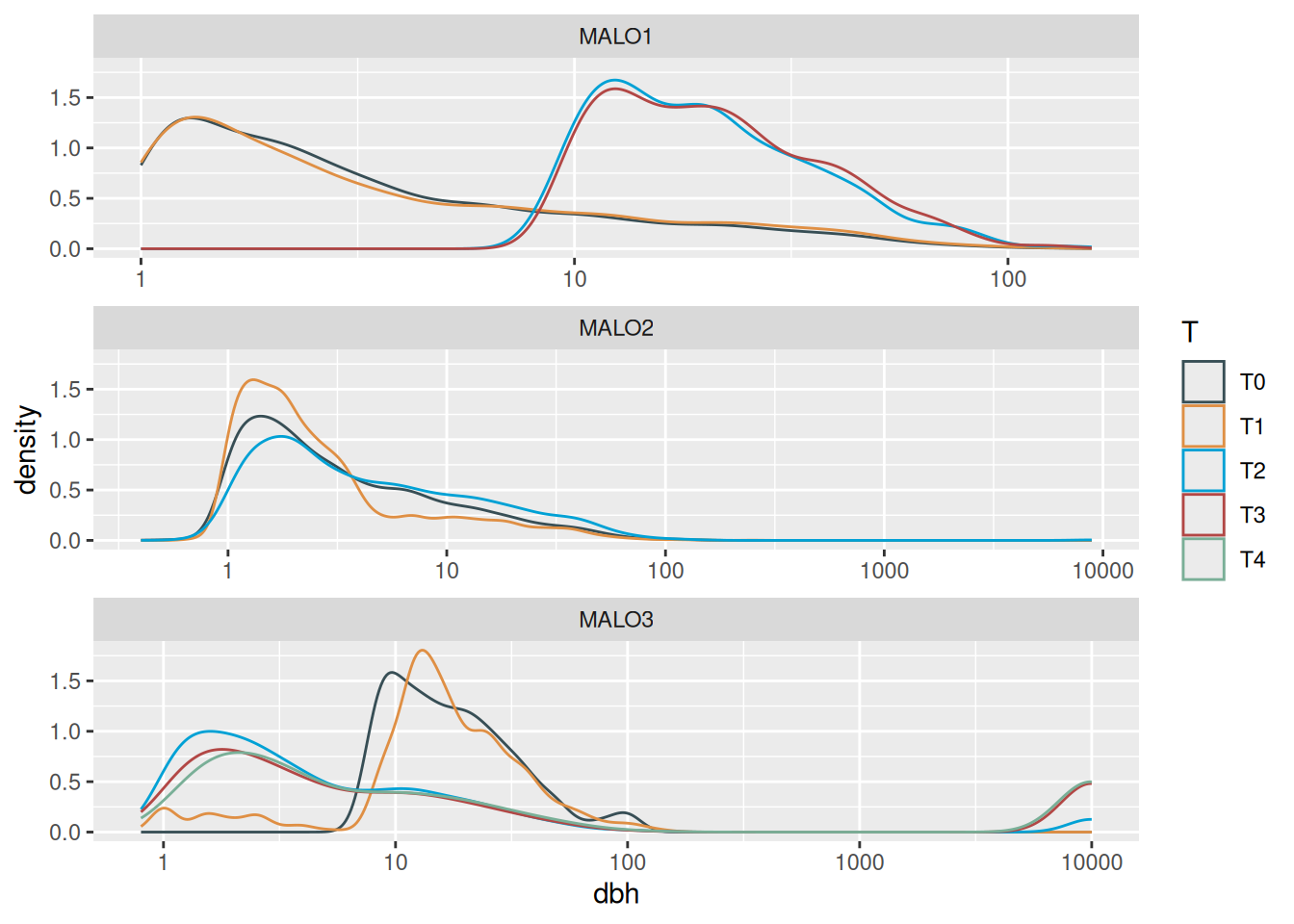
Warning in scale_y_log10(): log-10 transformation introduced infinite values.Warning: Removed 29810 rows containing non-finite outside the scale range
(`stat_ydensity()`).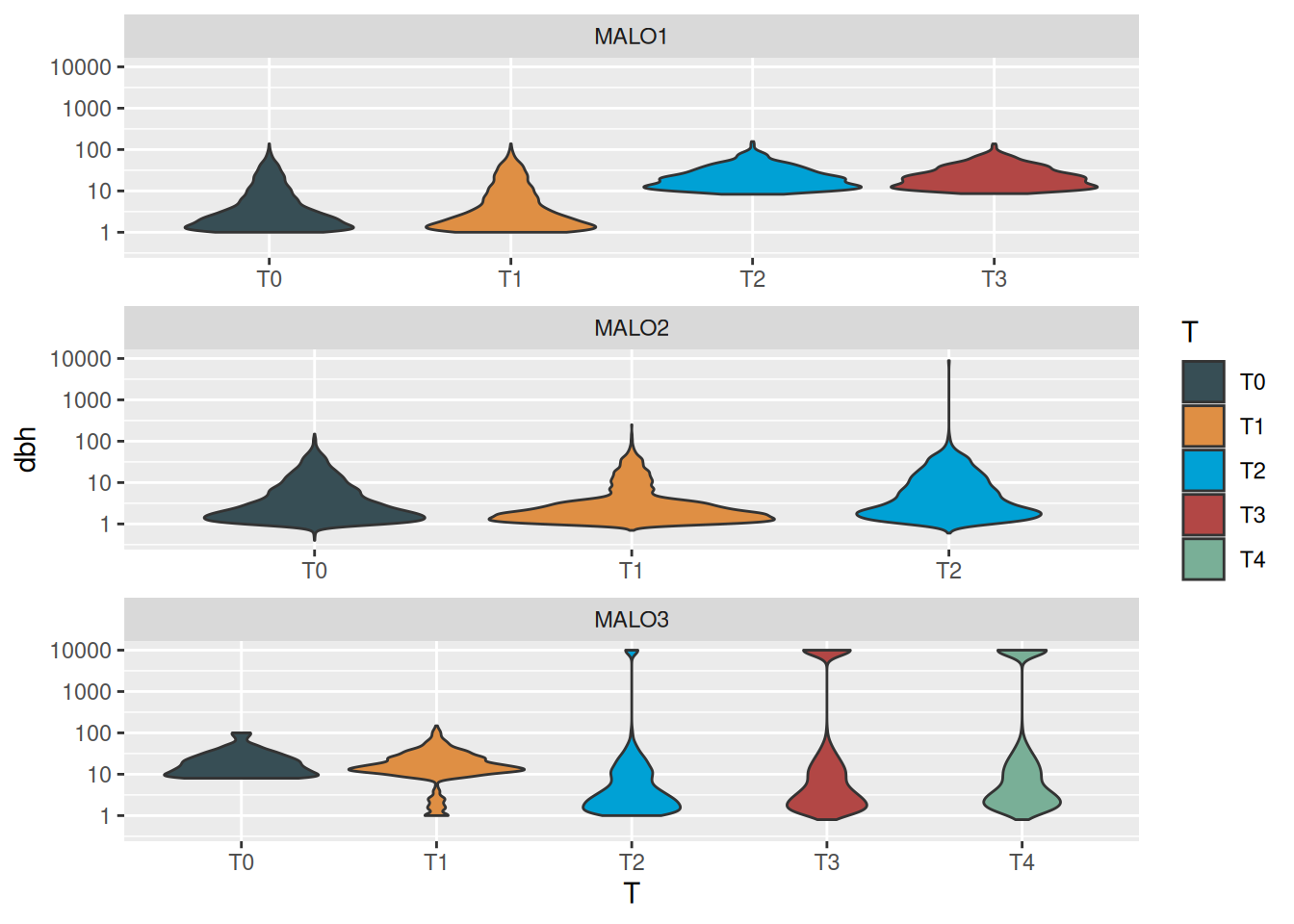
Annexes
Espèces présentes dans les parcelles à T3
| MALO1 | MALO3 | |
|---|---|---|
| ADEPAV | 0 | 1 |
| AGASAL | 1 | 1 |
| ALLBOR | 1 | 0 |
| ANTBOR | 1 | 1 |
| ANTMAD | 0 | 1 |
| ARTHET | 1 | 1 |
| BERBOR | 0 | 1 |
| BOEMAC | 0 | 1 |
| BRELAN | 0 | 1 |
| CALTAC | 1 | 1 |
| CASCOR | 1 | 1 |
| CASORI | 0 | 1 |
| CHACOR | 0 | 1 |
| CITAUR | 0 | 1 |
| CITRET | 0 | 1 |
| CLIHIR | 0 | 1 |
| CNEGLA | 0 | 1 |
| COFMAU | 1 | 1 |
| CYABOR | 1 | 0 |
| DIOBOR | 1 | 1 |
| DIODIG | 1 | 1 |
| DORAPE | 1 | 1 |
| DRAREF | 0 | 1 |
| DRYCAU | 1 | 0 |
| ERYLAU | 1 | 1 |
| FICDEN | 0 | 1 |
| FICLAT | 1 | 1 |
| FICMAU | 1 | 0 |
| FICREF | 1 | 1 |
| GAEVAG | 0 | 1 |
| HOMPAN | 1 | 1 |
| HYOIND | 0 | 1 |
| LABCAL | 1 | 1 |
| LEEGUI | 0 | 1 |
| LITCHI | 0 | 1 |
| LITGLU | 0 | 1 |
| MANIND | 0 | 1 |
| MEMCON | 0 | 1 |
| MEMCON? | 0 | 1 |
| MIMBAL | 1 | 1 |
| MOLALT | 1 | 1 |
| MYOOBO | 0 | 1 |
| NUXVER | 1 | 1 |
| OCHBOR | 1 | 0 |
| OCOOBT | 1 | 1 |
| PITSEN | 1 | 1 |
| POLAEM | 0 | 1 |
| POLREP | 1 | 0 |
| POUBOR | 0 | 1 |
| PSICAT | 0 | 1 |
| SCOHET | 0 | 1 |
| SIDBOR | 1 | 1 |
| SYZ | 0 | 1 |
| SYZBOR | 1 | 1 |
| SYZCUM | 0 | 1 |
| SYZCYM | 1 | 1 |
| SYZJAM | 0 | 1 |
| TABMAU | 1 | 1 |
| TAMELL | 1 | 1 |
| TREORI | 0 | 1 |
| XYLRIC | 1 | 1 |
Effectifs par espèce par parcelle à T3
| MALO1 | MALO3 | |
|---|---|---|
| ADEPAV | 0 | 1 |
| AGASAL | 20 | 4 |
| ALLBOR | 2 | 0 |
| ANTBOR | 152 | 734 |
| ANTMAD | 0 | 12 |
| ARTHET | 3 | 75 |
| BERBOR | 0 | 64 |
| BOEMAC | 0 | 3 |
| BRELAN | 0 | 19 |
| CALTAC | 8 | 7 |
| CASCOR | 8 | 5 |
| CASORI | 0 | 1 |
| CHACOR | 0 | 779 |
| CITAUR | 0 | 6 |
| CITRET | 0 | 1 |
| CLIHIR | 0 | 6 |
| CNEGLA | 0 | 2 |
| COFMAU | 1 | 5 |
| CYABOR | 3 | 0 |
| DIOBOR | 22 | 19 |
| DIODIG | 17 | 7 |
| DORAPE | 37 | 458 |
| DRAREF | 0 | 13 |
| DRYCAU | 1 | 0 |
| ERYLAU | 2 | 148 |
| FICDEN | 0 | 5 |
| FICLAT | 8 | 17 |
| FICMAU | 9 | 0 |
| FICREF | 1 | 12 |
| GAEVAG | 0 | 16 |
| HOMPAN | 47 | 68 |
| HYOIND | 0 | 45 |
| LABCAL | 240 | 755 |
| LEEGUI | 0 | 1 |
| LITCHI | 0 | 3 |
| LITGLU | 0 | 16 |
| MANIND | 0 | 18 |
| MEMCON | 0 | 128 |
| MEMCON? | 0 | 1 |
| MIMBAL | 2 | 246 |
| MOLALT | 12 | 16 |
| MYOOBO | 0 | 31 |
| NUXVER | 64 | 12 |
| OCHBOR | 1 | 0 |
| OCOOBT | 13 | 30 |
| PITSEN | 1 | 19 |
| POLAEM | 0 | 1 |
| POLREP | 12 | 0 |
| POUBOR | 0 | 3 |
| PSICAT | 0 | 1 |
| SCOHET | 0 | 1 |
| SIDBOR | 3 | 30 |
| SYZ | 0 | 1 |
| SYZBOR | 14 | 59 |
| SYZCUM | 0 | 2 |
| SYZCYM | 21 | 75 |
| SYZJAM | 0 | 6 |
| TABMAU | 2 | 12 |
| TAMELL | 15 | 74 |
| TREORI | 0 | 1 |
| XYLRIC | 18 | 1 |
Warning in attr(x, "align"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")Warning in attr(x, "format"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")Warning in attr(x, "align"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")Warning in attr(x, "format"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")Warning in attr(x, "align"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")Warning in attr(x, "format"): 'xfun::attr()' est obsolète.
Utilisez plutôt ‘xfun::attr2()’.
Voir help("Deprecated")